Você herdou genes de seus pais, mas seu material genético não é um roteiro fixo. O DNA contém a informação, mas são as marcas epigenéticas — como acetilação, metilação, fosforilação e glicanos adicionados às histonas e ao DNA — que dizem quais genes estão ligados, desligados ou em espera.
O metabolismo da célula — ou seja, a forma como usamos glicose, aminoácidos e lipídios para gerar energia — fornece diretamente os cofatores e substratos usados pelas enzimas epigenéticas. Em outras palavras: os nutrientes que ingerimos e o estado energético da célula se traduzem em marcas químicas que modulam a expressão gênica.
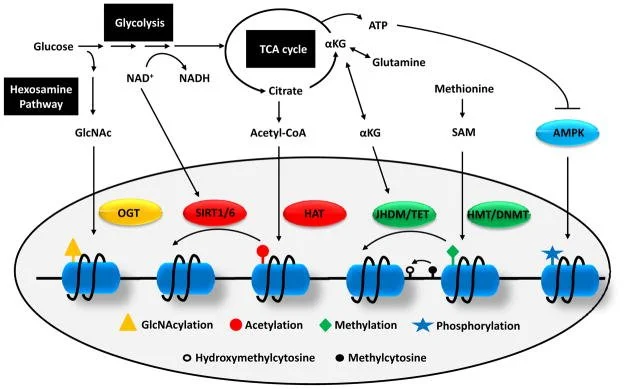
Glicose, aminoácidos e o estado energético geram metabólitos-chaves (NAD⁺, acetil-CoA, α-cetoglutarato, SAM, UDP-GlcNAc, ATP) que atuam como substratos ou cofatores de enzimas epigenéticas — por exemplo, sirtuínas, HATs, Jmj/TET, HMT/DNMT, OGT e AMPK. Essas enzimas colocam ou retiram marcas químicas nas histonas (acetilação, metilação, fosforilação, O-GlcNAc) e no DNA (5mC, 5hmC), alterando a acessibilidade da cromatina e, portanto, quais genes são ligados ou desligados.
Glicólise / NAD⁺ ⇄ NADH → Sirtuínas (SIRT1/6)
O estado redox (relação NAD⁺/NADH) controla sirtuínas, desacetilases dependentes de NAD⁺. Mais NAD⁺ → maior atividade das sirtuínas → remoção de acetilos em histonas e fatores transcricionais → geralmente repressão de programas anabólicos e ativação de vias de economia/reciclagem (ex.: autofagia).
Ciclo do TCA → Citrato → Acetil-CoA → HATs (acetilação das histonas)
O citrato exportado para citosol/núcleo vira acetil-CoA (via ATP-citrato liase). Acetil-CoA é o doador de grupos acetil: HATs usam-no para acetilar lisinas de histonas, relaxando cromatina e favorecendo transcrição. Alterações na disponibilidade de acetil-CoA linkam estado nutricional ao nível global de acetilação.
Glutamina → α-cetoglutarato (αKG) → Jmj/TET (desmetilases dependentes de αKG)
αKG é cofator para as dióxigenases da família Jumonji (remoção de metilas em histonas) e para as enzimas TET (oxidação da 5mC para 5hmC no DNA). A presença de αKG facilita desmetilação; por outro lado, acúmulos de metabólitos análogos (ex.: 2-HG, succinato, fumarato) bloqueiam essas enzimas.
Metionina → SAM (S-adenosilmetionina) → HMTs / DNMTs (transferência de metilas)
SAM é o doador universal de grupos metila. HMTs metilam histonas; DNMTs metilam DNA (5mC). Assim, o estado do ciclo de um-carbono (folato/metionina) regula a metilação epigenética.
Via das hexosaminas → UDP-GlcNAc → OGT (O-GlcNAcilação)
Uma fração da glicose entra na via das hexosaminas formando UDP-GlcNAc. A OGT transfere GlcNAc para serinas/treoninas de proteínas nucleares (incluindo histonas e co-reguladores), modulando função e interações proteicas.
ATP / AMP → AMPK → fosforilação de histonas/enZimas
Em baixa energia (AMP↑), AMPK ativa-se e pode fosforilar histonas ou enzimas epigenéticas, promovendo respostas transcricionais ao estresse energético.
Resumindo, nutrição e energia são traduzidas em ‘marcas’ epigenéticas que reprogramam programas transcricionais — com impacto em diferenciação, resposta ao estresse, envelhecimento e câncer.
Por que isso importa?
Estes processos têm algumas implicações biológicas e clínicas:
Plasticidade transcricional: permite que células ajustem programas gênicos de forma rápida e reversível conforme nutrientes/energia.
Memória epigenética: mudanças sustentadas (p.ex. metilação do DNA) podem preservar estados celulares induzidos por ambiente/nutrição.
Doença e terapia: muitos tumores e distúrbios metabólicos exibem alterações epigenéticas causadas por reprogramação metabólica — e algumas terapias visam esses pontos (inibidores de DNMTs, moduladores de SIRT, etc.).
Intervenções (nutrição/estilo de vida): evidências experimentais sugerem que dieta, jejum intermitente e exercício alteram metabólitos epigenéticos; contudo, efeitos em humanos são contextuais e dependem de tecido, dose e duração — cuidado com extrapolações simplistas.